产品介绍
ATAC-seq(Assay for Transposase Accessible Chromatin with high throughput sequencing)该技术通过改造后活性极高的转座酶介导,对染色质结构开放区域进行捕获测序,仅需少量细胞便可获得实时全基因组活性调控序列信息。该技术通过转座酶对某种特定时空下开放的核染色质区域进行切割,进而获得在该特定时空下基因组中所有活跃转录的调控序列。
真核生物的DNA并不是裸露的,而是被包装成核小体形成串珠状结构并进一步被折叠、包装。而基因的转录,需要将这种高级结构解开,使DNA成为可以使各种转录机器与其结合的裸露状态,即形成开放染色质区域。ATAC-Seq作为一种新型的研究开放染色质的技术,利用Tn5转座酶进入并切割裸露的DNA,并同时连接上特异性的测序接头。因为切割和加接头一步完成,因此该技术可大大降低所需细胞起始量。
技术优势
1.所需细胞起始量低
- 应用范围广,适用于大部分物种及细胞类型。
实验方案

信息分析
技术参数
| 样本要求 | 项目周期 |
| 取生长状态良好的细胞进行胰蛋白酶消化计数,保证细胞活率在85%以上;将细胞重悬在冻存液中,每个冻存管加入8~10万细胞(1ml冻存液即可。细胞数量不要太多,也不要太少,尽量在这个范围,否则效果都会有影响);将冻存管放入程序降温盒中直接放入-80度冰箱即可注意:1.建议使用程序降温盒,一是可以减少操作步骤,二是效果要比传统的异丙醇要好,可以跟试剂商购买。 2.冻存液基本配方:50% FBS+40%完全培养基+10% DMSO.(DMSO浓度不要高于这个,否则会损坏细胞,导致死亡率增高) 3.运输保证足够干冰,将细胞放入中间,避免增温导致细胞死亡。 | 15个样本以下标准流程的运转周期约为50个工作日。样品数较多时,项目周期需要与技术支持人员确定。 |
案例分享
案例:
ATAC-Seq分析显示年龄相关性黄斑变性患者的染色质可及性普遍降低
Wang J, Zibetti C, Shang P, et al. ATAC-Seq analysis reveals a widespread decrease of chromatin accessibility in age-related macular degeneration[J]. Nature Communications, 2018, 9(1).
背景:
年龄相关性黄斑变性(AMD)是导致老年人视力丧失的主要因素,大多发生于45岁以上,其患病率随年龄增长而增高。目前AMD进程中表观遗传的改变尚不清楚。本文利用ATAC-Seq对AMD患者视网膜(retina)及视网膜色素上皮细胞(RPE)进行染色质易近性进行了研究。
方法:
利用ATAC-Seq技术鉴定AMD及正常组织的retina及RPE的开放染色质区域。另外,结合RNA-Seq技术对开放染色质区域与基因表达的关系进行分析。
结果:
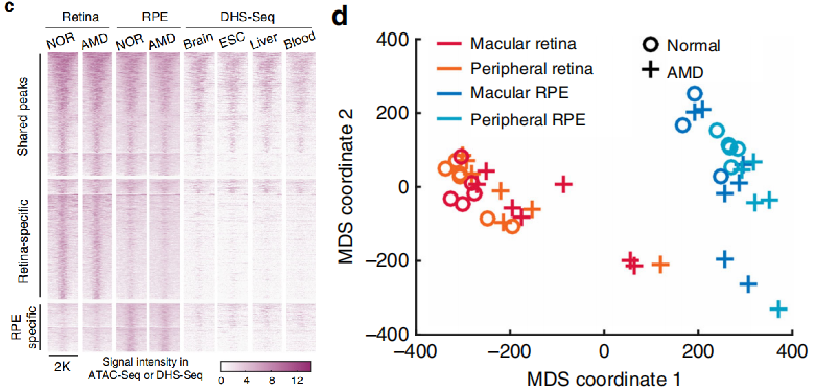
将鉴定出的peaks(开放染色质区域)分类得到retina特异、RPE特异及两者共有的peaks,与不同组织比较(DNase-Seq数据),发现两者共有的peaks大部分是组织特异的,retina及RPE特异的peaks大多是组织特异的(左图)。利用peaks将样本分类,发现retina和RPE明显分开,正常组织和AMD也有分离的趋势(下图)。
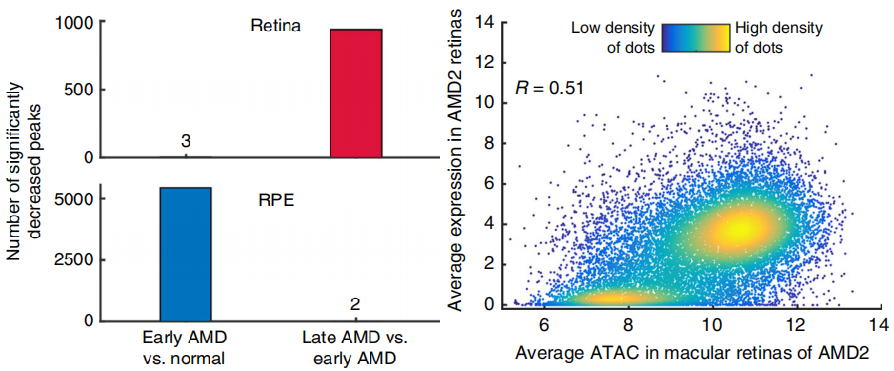
分别鉴定retina和RPE AMD早期 vs 正常组织以及AMD晚期 vs AMD早期的差异开放染色质区域(AMD),表明retina染色质易近性主要在AMD早期到AMD晚期出现下降,而RPE染色质易近性主要在正常组织到AMD早期出现下降(下左图)。结合转录组分析表明,ATAC-Seq peaks强度和基因表达呈现较强相关性,相关系数为0.51(下右图)。