产品介绍
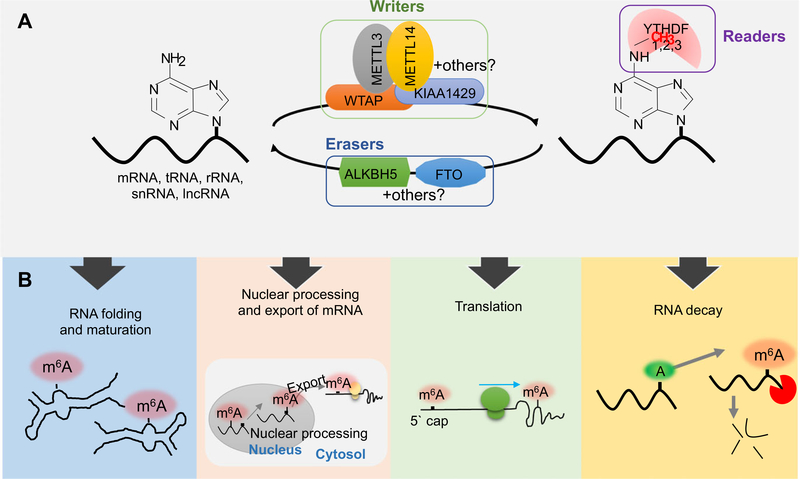
m6A甲基化修饰是真核生物mRNA甚至病毒RNA广泛存在的一种RNA修饰形式。2011年,第一个真正意义上的RNA去甲基化酶FTO的报道,使得mRNA/lncRNA的甲基化研究再次进入人们的视野。MeRIP通过m6A特异性抗体富集和测序,用于研究RNA的腺苷甲基化修饰。
功能简介
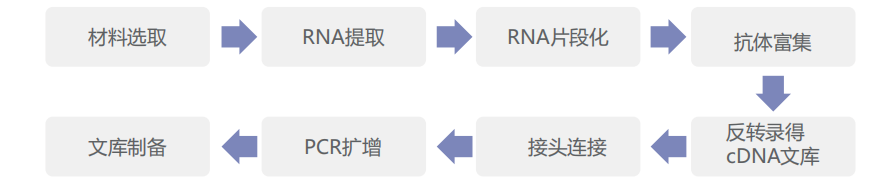
实验策略
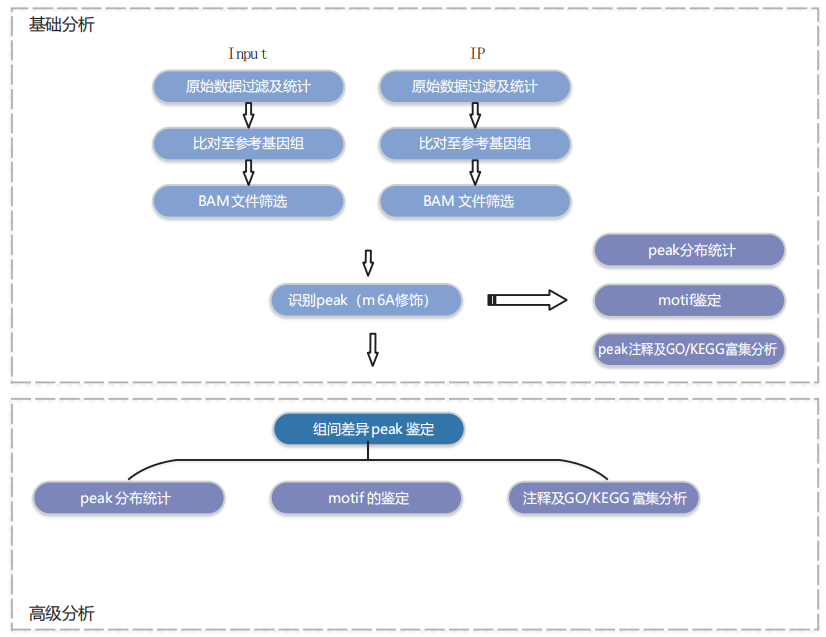
信息分析
技术参数
| 样本要求 | 项目周期 |
| 总量:不同样本需求不同,人类样本≥ 2μg;样本类型:去蛋白并进行DNase处理后的完整总RNA样本总量:常规样本建议不少于70ug,微量方法最低可到8ug样本浓度:建议≥70 ng/μL样本完整度:RIN ≥ 7,Ratio 28S/18S ≥ 0.7;某些来源样本(如体液样本,昆虫样本,水生生物样本等)无RIN和Ratio28S/18S要求 | 15个样本以下标准流程的运转周期约为50个工作日。样品数较多时,项目周期需要与技术支持人员确定。 |
案例分析
案例:
MeRIP-seq和RNA-seq联合分析研究热休克反应的分子机制
Dynamic m6A mRNA methylation directs translational control of heat shock response
背景:
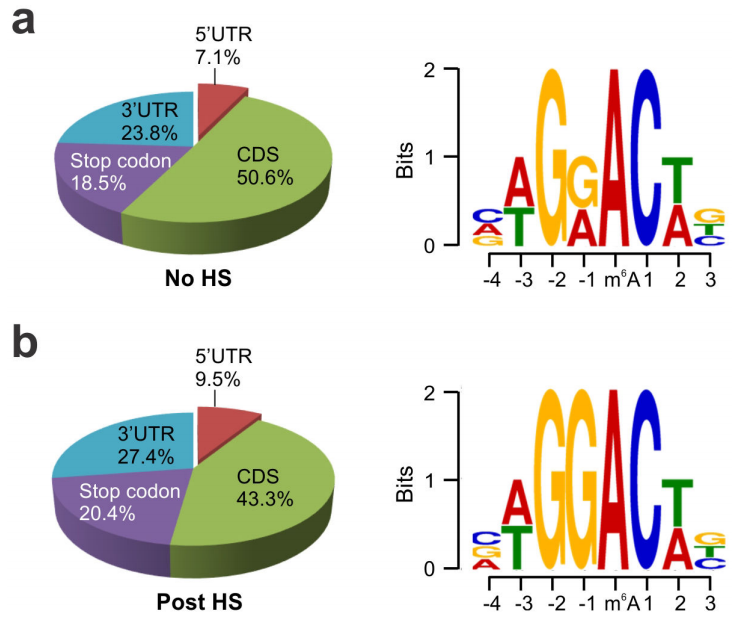
热休克反应是机体在热应激情况下,表现出的以基因表达变化为特征的防御适应性反应,是生物体面对刺激时的非特异性应急反应,以保证细胞短暂适应极端环境。热休克蛋白(heat shock protein, HSP)在其中起着重要的作用。m6A是最丰富的mRNA表观修饰,调节转录本的可变剪切、RNA与蛋白质的相互作用以及RNA的稳定性。在哺乳动物细胞中,m6A并不是对称分布,在mRNA的5’UTR甲基化水平比其他功能区域低。然而,人们对5’UTR甲基化的调控基因表达机制知之甚少,且5’UTR在翻译的起始过程中起着至关重要的作用,但是对m6A在翻译中的调控作用所知甚少。
方法:

结果:
研究了m6A与热休克反应的关系,绘制出了热处理影响热诱导mRNA的5’UTR甲基化水平变化的分子机制:核定位的m6A识别蛋白YTHDF2抑制m6A去甲基化蛋白FTO与RNA的5’UTR区相互作用,从而保留热诱导RNA的5’UTR甲基化位点。研究热诱导RNA 5’UTR甲基化的动态变化与热休克反应的关系,不仅扩大了m6A调控广度,而且揭示了一个未知的热休克反应的转录后调控机制,为我们研究表观修饰提供了新角度。