产品介绍
目标区域捕获重亚硫酸盐甲基化测序是通过设计相应的探针序列,特异性捕获感兴趣的基因组特定区域,捕获片段进一步通过重亚硫酸盐处理,经高通量测序检测目标区域的甲基化水平。该技术是大样本量,固定区域甲基化检测的首选,可广泛应用于临床样本甲基化特异性区域的鉴定以及与疾病相关的甲基化Bio-Marker的筛选和验证。
技术优势
1.覆盖位点多
获得比甲基化芯片更多的CpG位点
2.检测范围广
除了覆盖已知的DMR区域之外,能发现新的DMR区域
3.精确度高
精确到单碱基分辨率,与已发表的全基因组甲基化测序数据有高度一致性
4.精确度高
目标区域富集、测序深度分布、覆盖度的重复性高;优先选用AgilentSureSelectXT Human Methyl-Seq 捕获探针,目标区域包括:
① 目标区域为84M,检测3.7M的CpG位点;
② 主要针对CpG island、Gencode promoter和已知调控区域;
③ 覆盖已知的癌症特异DMR区域和组织特异性DMR区域;
④ 包含CpG 岛上下游 4kb范围的“shores”和 “shelves”区域;
⑤ 包含脱氧核糖核酸酶I(DNase I)高敏感性位点。也可以采用全基因组编码基因启动子芯片(TSS上游2k,下游500bp,NimbleGene 芯片)。
实验策略

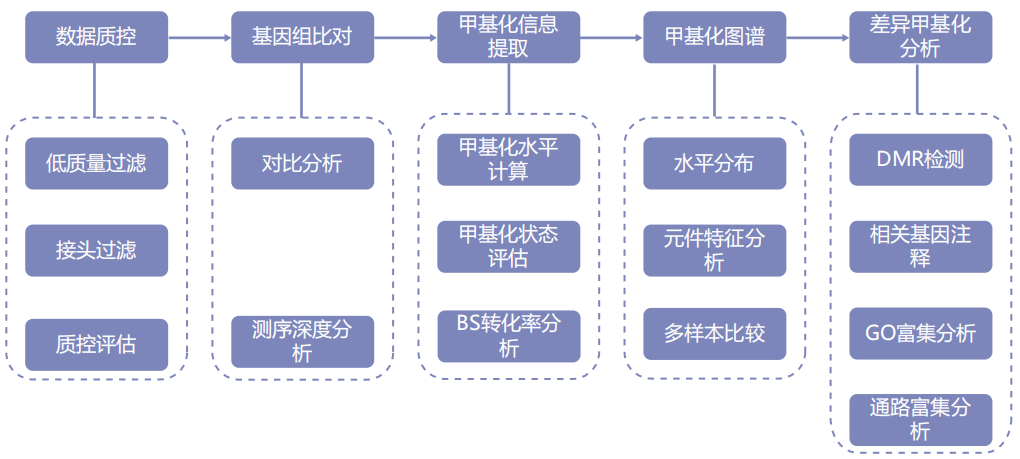
信息分析
技术参数
| 样本要求 | 项目周期 |
| 总量:不同样本需求不同,人类样本≥ 2μg;纯度: OD260/280 = 1.8~2.0;完整性:DNA无明显降解,需提供凝胶电泳检测胶图。可提供标准芯片: ①全基因组编码基因启动子芯片(TSS上游2k,下游500bp,NimbleGene 芯片)②Agilent SureSelectXT Human Methyl-Seq芯片 | 15个样本以下标准流程的运转周期约为50个工作日。样品数较多时,项目周期需要与技术支持人员确定。 |
案例分析
案例:
基于液相杂交捕获的重亚硫酸盐测序的肝细胞癌DNA甲基化研究
Gao, F., et al. (2015).Global analysis of DNA methylation in hepatocellular carcinoma by a liquid hybridization capture-based bisulfite sequencing aproach. Clin Epigenetics 7: 86.
背景:
全基因组重亚硫酸盐甲基化测序的测序成本较高,DNA甲基化的修饰研究往往只关注基因附近的区域,对基因组的区域进行选择性的捕获和重亚硫酸盐测序可以有效降低测序成本,并期望将其运用于癌症表观基因组学研究领域。
方法:
基于目标区域液相捕获的重亚硫酸盐测序技术,对肝细胞癌的启动子区DNA甲基化展开研究。
结论:
建立了基于全基因组启动子区域液相杂交捕获和重亚硫酸盐测序相结合的技术,并利用该技术研究了8个肝细胞癌患者癌和癌旁组织的全基因组启动子区域的DNA甲基化水平,共涉及到1.86Mb的CpG位点。同时,结合基因表达数据和后期的大规模样本间验证,成功鉴定出了7个与肝癌发生和发展有关的差异甲基化基因。
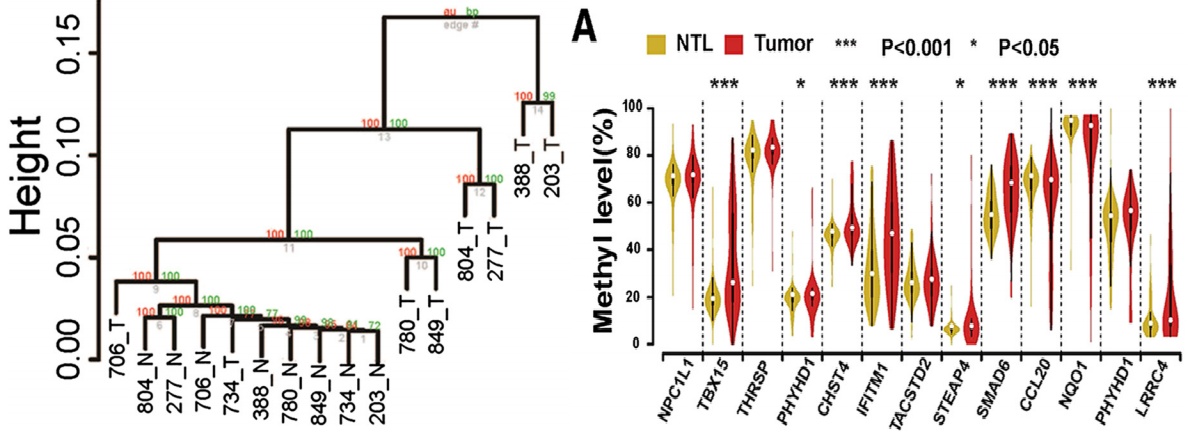
8对肝细胞癌患者癌和癌旁组织的DNA甲基化聚类分析以及差异甲基化候选基因


